1、引物设计原则
lncRNA:①引物应在核酸序列保守区内设计并具有特异性;②引物长度一般在18-25碱基之间;③避免连续出现相同的核苷酸;④CG含量在45%-55%之间;⑤上下游引物Tm值差控制在5℃以内;⑥扩增产物长度在80-250bp。最长不要超过300bp;⑦引物内部或引物之间避免形成二级结构、发夹结构。
miRNA:①引物与模板的序列要紧密互补;②引物不能在模板的非目的位点引发DNA聚合反应;③引物与引物之间应避免形成稳定的二聚体或发夹结构;④引物的长度一般为15-30bp,常用的是18-27bp,但不应大于38bp,因为过长会导致其延伸温度大于74℃,不适于TaqDNA聚合酶进行反应;⑤引物序列的GC含量一般为40-60%,过高或过低都不利于引发反应,且上下游引物的GC含量不能相差太大。
circRNA:①对于外显子环化circRNA,引物跨剪切位点(backsplice)设计;②对于内含子环化circRNA,可跨剪切位点设计,也可围绕内含子区域设计引物;③扩增产物长度建议不超过100bp。
2、引物设计流程
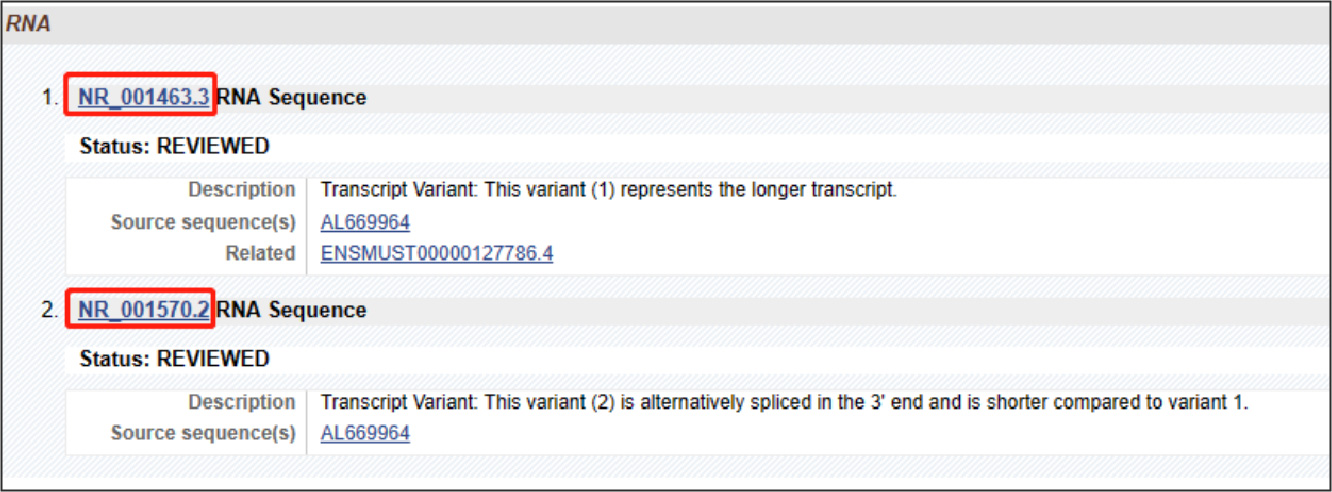
lncRNA:在NCBI等数据库中查找目的基因序列(NCBI通常以NR开头)
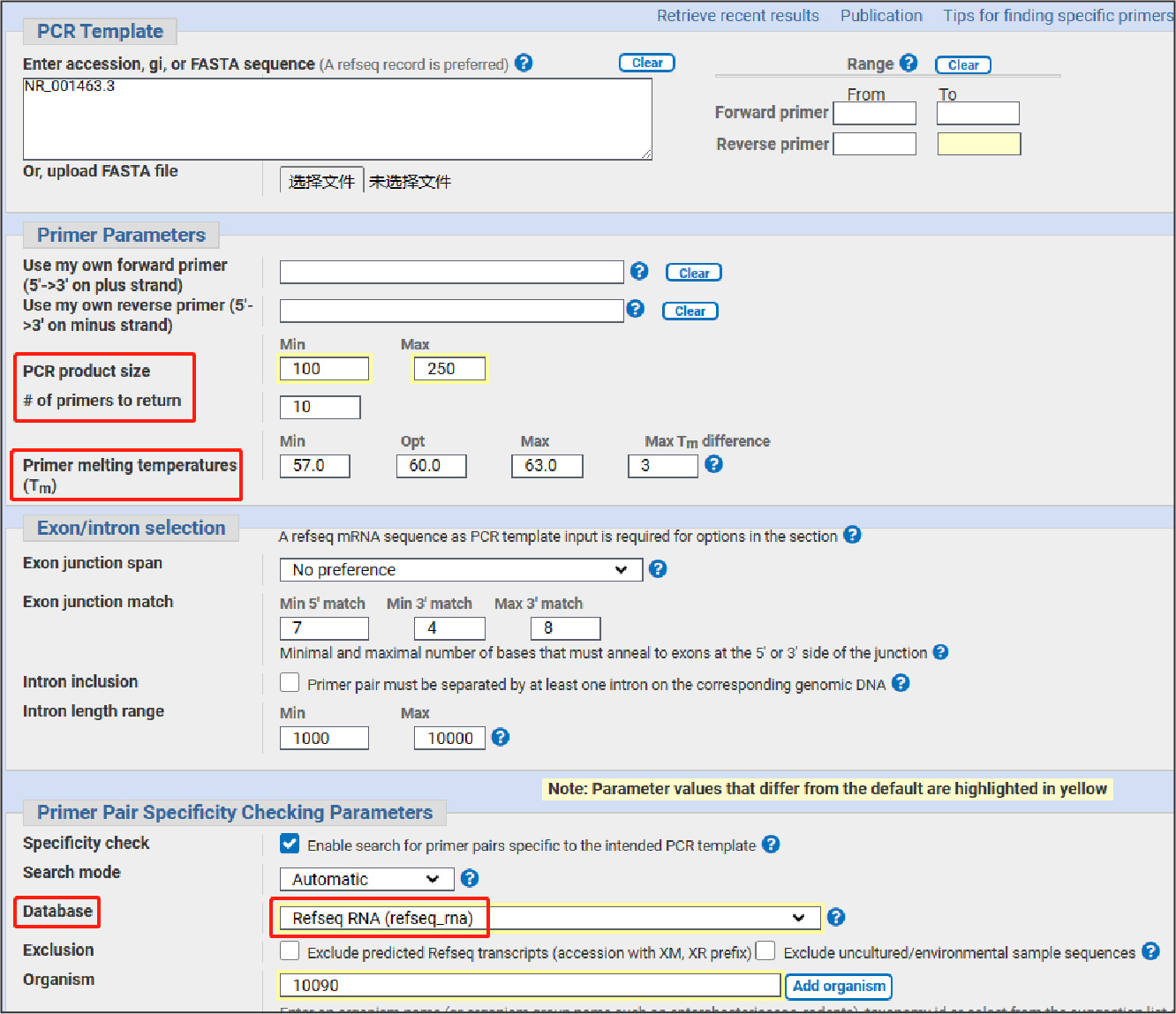
进入序列信息页,点击右侧的“pick primers”:
遵循引物设计原则填写相关信息,开始检索:
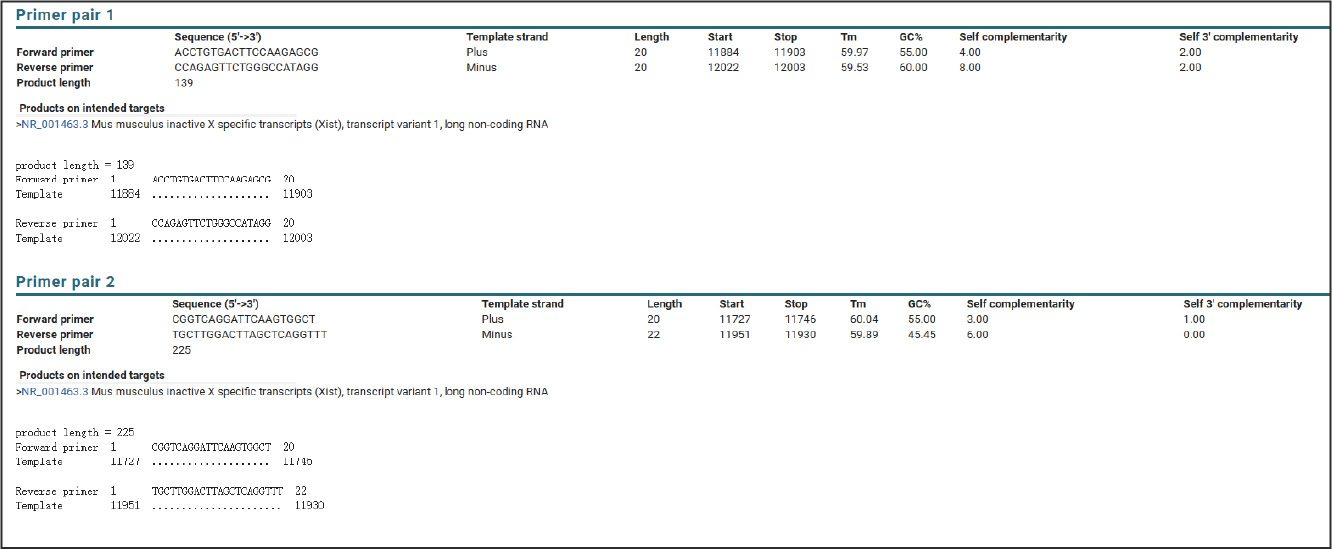
选择特异性较高的引物进行后续实验:
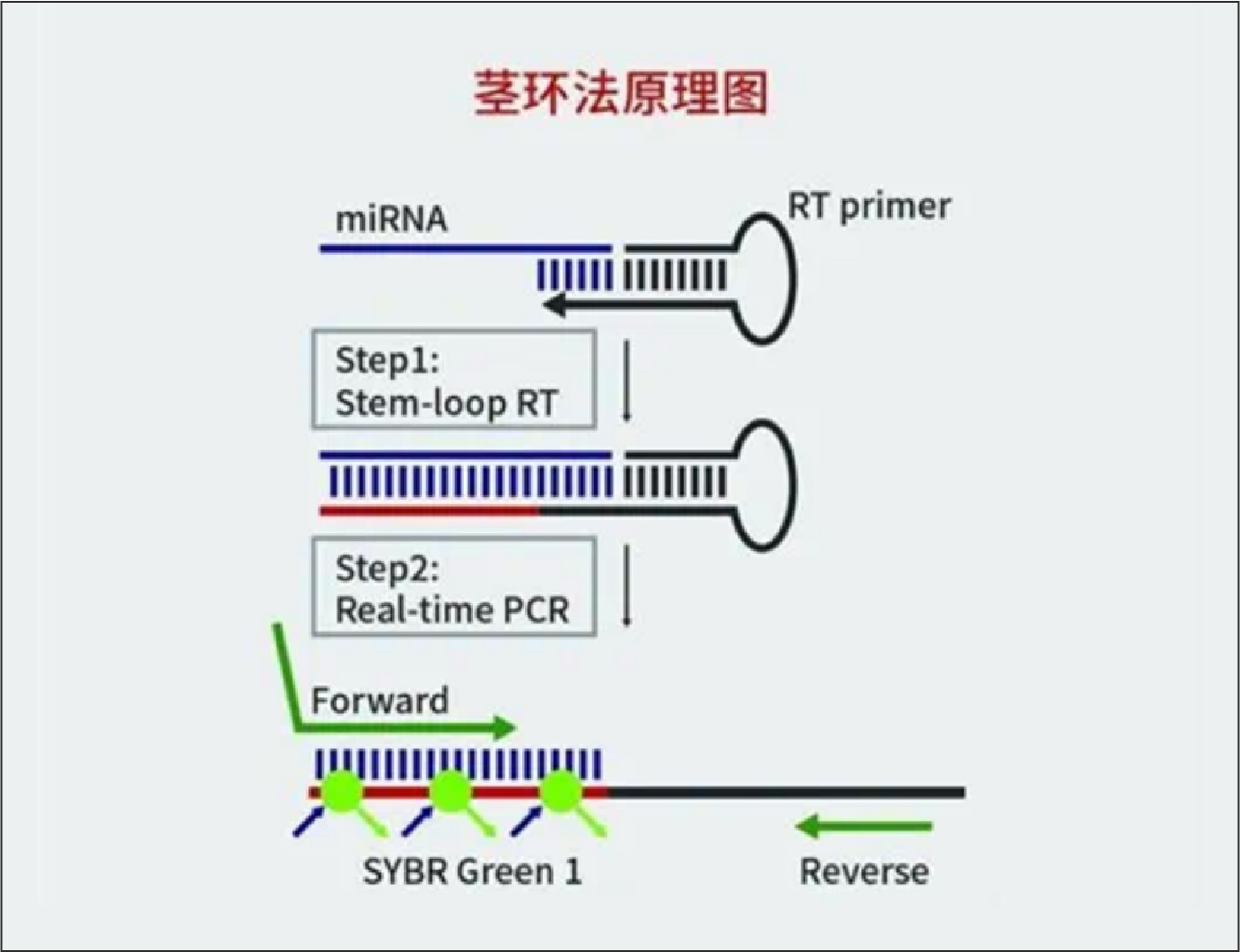
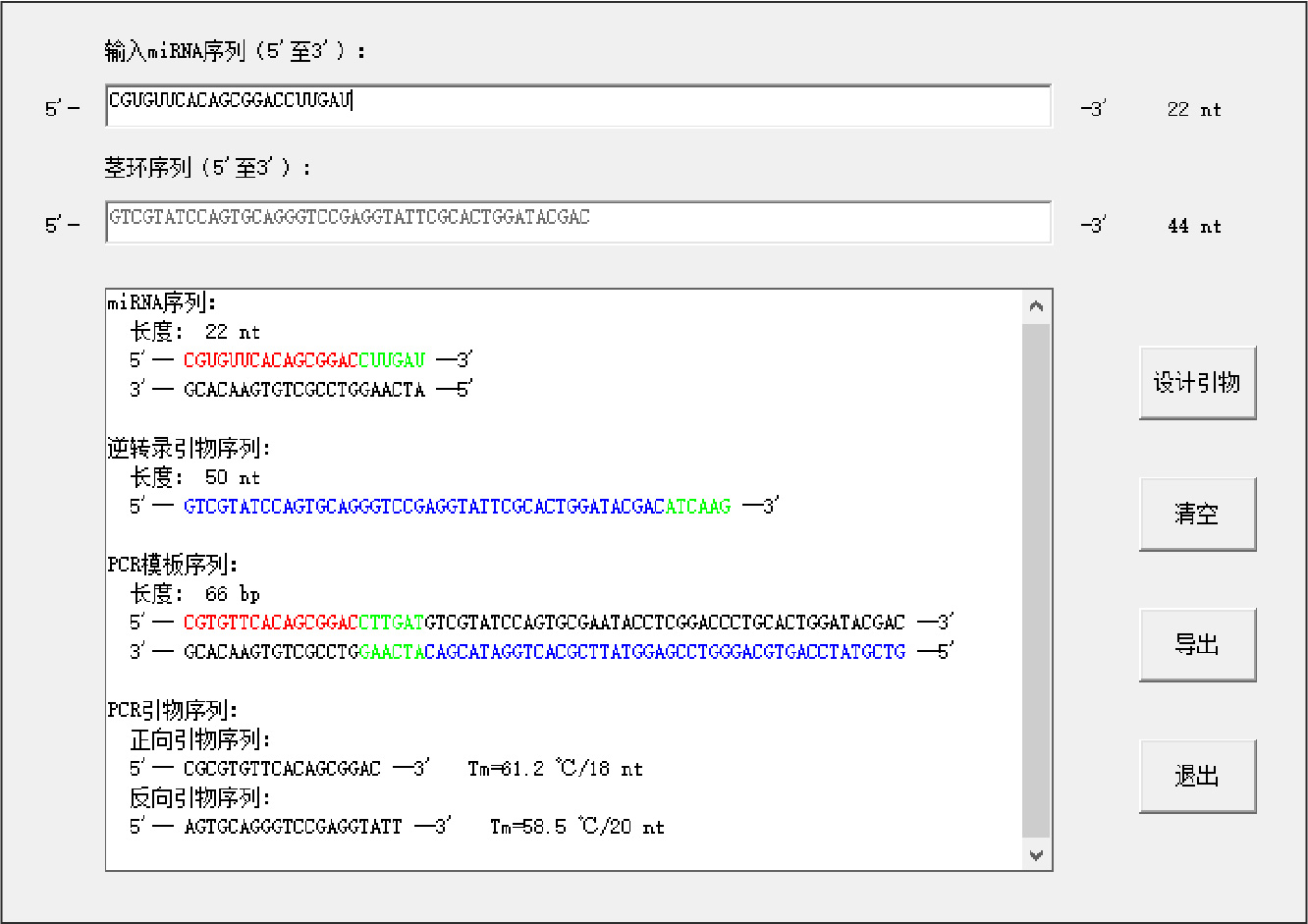
miRNA:在miRBase等数据库中查找目的基因序列,采用茎环法设计反转录引物和用于qPCR检测的引物。
①反转录引物:通用的茎环序列+5~8个与目的miRNA的3’端反向互补的碱基;
②qPCR检测正向引物:根据miRNA的序列设计,一般用除去3’端6个碱基的剩余部分。若GC含量较低,可在5’端增加G/C进行调增,使引物Tm值接近60℃;③qPCR检测反向引物:为通用引物,一般选取茎环结构中的一部分。
经典茎环序列为:5'-GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGAC-3'
以mmu-miR-124-5p为例设计引物,其成熟序列为CGUGUUCACAGCGGACCUUGAU,选择使用miRNA引物设计软件“miRNA Design”获得引物序列。
circRNA:在circBase数据库中找到目的基因序列,circRNA全长序列的尾部和头部各选取150-300bp,将3’端序列放置在5’序列之前,获得新的序列,将该序列导入引物设计软件(Primer Premier系列软件)进行引物设计(可背靠背或跨剪切位点进行设计)。设计好的上游引物序列在原始序列的尾部,下游引物在原始序列的头部。确保扩增产物包含剪切位点!
以mmu_circ_0000151为例设计引物,其基因序列为,绿色代表序列前端,蓝色代表序列后端:
经过处理后得到的新序列为:
将该序列引物设计软件(Primer Premier系列软件)进行引物设计。
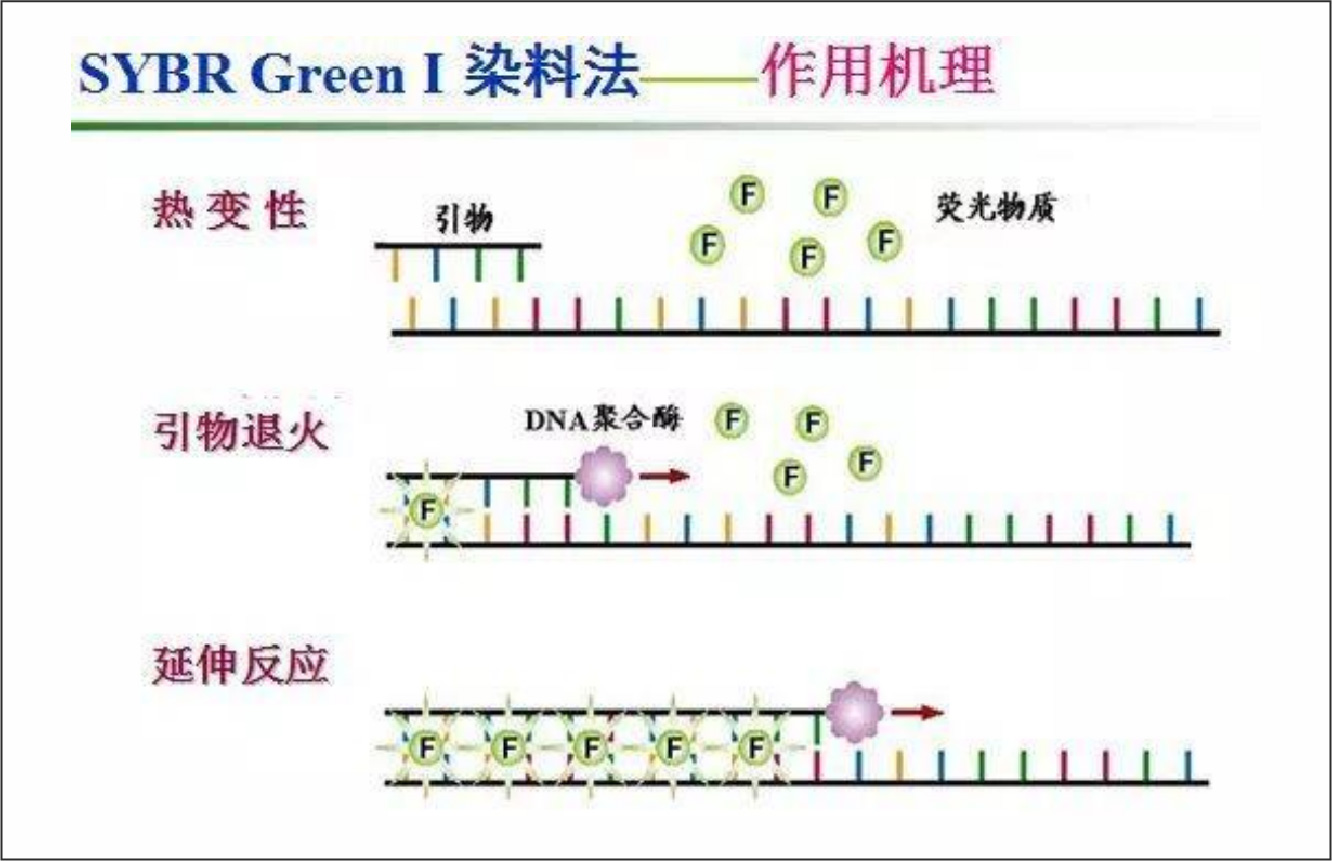
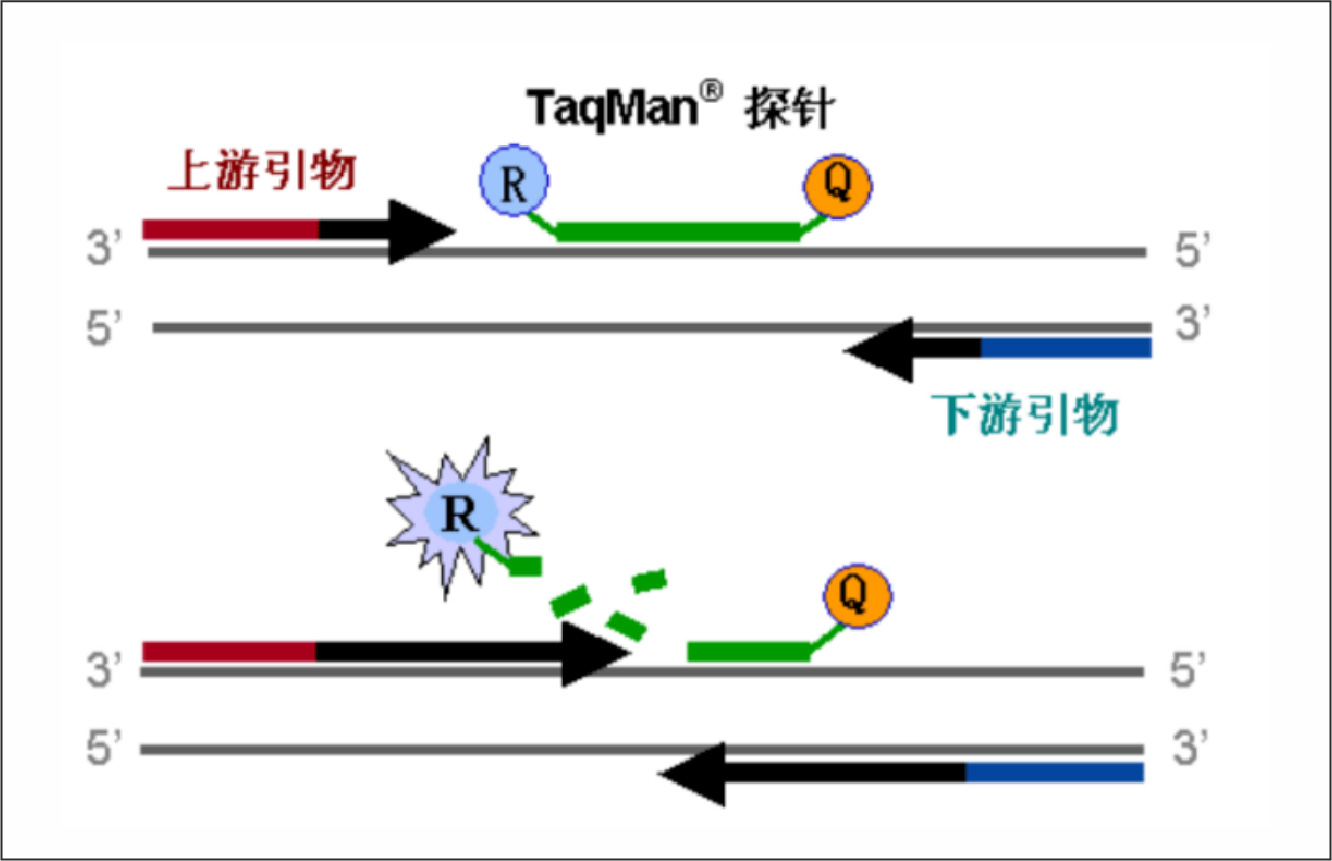
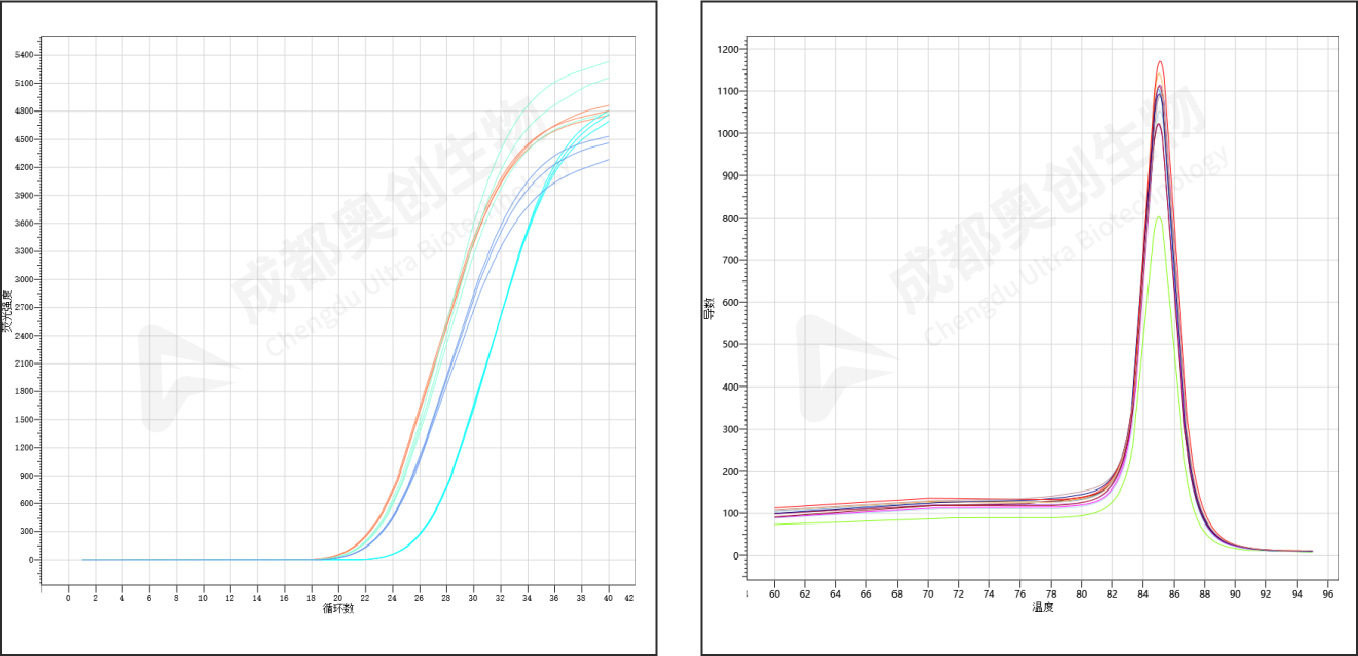
引物设计后需要通过观察溶解曲线的峰形是否为单峰来判断引物是否具有特异性,更多引物设计相关知识可以联系奥创生物技术负责人沟通讨论